高性價比的CALB固定化酶
- 分類:行業動態
- 發布時間:2024-09-06 09:22
- 訪問量:
【概要描述】1988年,科學家從(cong) 極端的南極環境中分離到了南極假絲(si) 酵母(Candida antarctica),這類酵母可以產(chan) 生兩(liang) 種脂肪酶,分別為(wei) 南極假絲(si) 酵母脂肪酶A和南極假絲(si) 酵母脂肪酶B
高性價比的CALB固定化酶
【概要描述】1988年,科學家從(cong) 極端的南極環境中分離到了南極假絲(si) 酵母(Candida antarctica),這類酵母可以產(chan) 生兩(liang) 種脂肪酶,分別為(wei) 南極假絲(si) 酵母脂肪酶A和南極假絲(si) 酵母脂肪酶B
- 分類:行業動態
- 發布時間:2024-09-06 09:22
- 訪問量:
一、CALB簡介
1988年,科學家從(cong) 極端的南極環境中分離到了南極假絲(si) 酵母(Candida antarctica),這類酵母可以產(chan) 生兩(liang) 種脂肪酶,分別為(wei) 南極假絲(si) 酵母脂肪酶A和南極假絲(si) 酵母脂肪酶B[1]。CALB是來源南極假絲(si) 酵母的脂肪酶B,在畢赤酵母中發酵表達得到。該酶能在水相或有機相中高效催化酯水解、酯的合成、酯交換、內(nei) 酯開環聚合、胺解反應、酰胺的水解與(yu) 合成及加成反應等,具有較強的手性選擇性及位置選擇性,廣泛用於(yu) 油脂加工、食品、醫藥、日化等行業(ye) 。
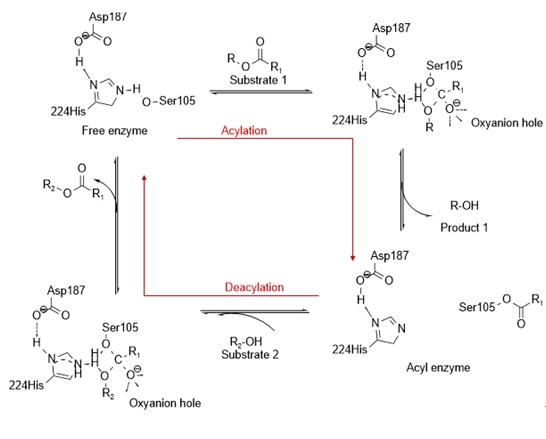
1994年,Uppenberg首次確定了CALB的序列及晶體(ti) 結構,CALB由317個(ge) 氨基酸組成,是一類具有α/β水解酶超家族折疊結構的絲(si) 氨酸水解酶[2]。CALB的活性中心由催化三聯體(ti) (Ser105-Asp187-His224),氧負離子洞和底物結合口袋組成。Ser105 位於(yu) β4 股和α4 螺旋的轉角處,稱為(wei) “親(qin) 核肘”(nucleophilic elbow)。通常,在α/β水解酶折疊家族中,Ser附近最保守的序列區域是GXSXG,但在CALB 中,該序列為(wei) TWSQG,保守序列的變化造成“親(qin) 核肘”的轉角處變寬。His224 位於(yu) α9螺旋前端轉角處,並指向活性位點。Asp187位於(yu) β6股後的轉角處,其側(ce) 鏈氧原子與(yu) His224 之間形成氫鍵[2,3]。
實例 1(胺解反應)[4]
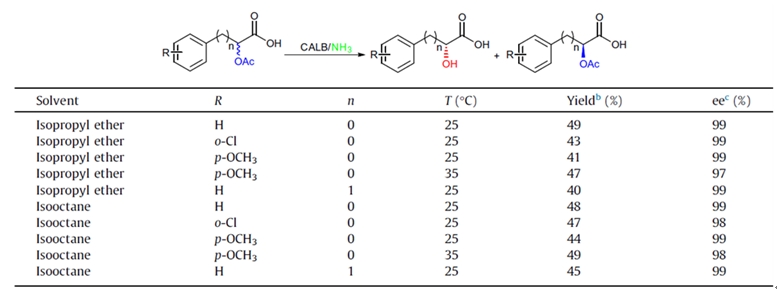
實例 2(胺解反應-手性拆分)[5]

實例 3(內(nei) 酯開環聚合反)[6]

實例 4(酯交換反應-羥基位置選擇性)[7]

實例 5(酯交換反應-醇的手性拆分)[8]

實例 6(酯合成反應-酸的手性拆分)[9]
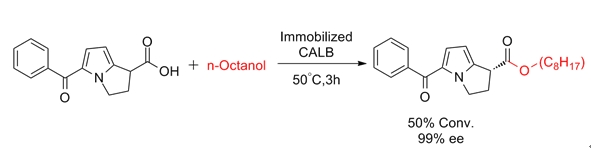
實例 7 (酯水解反應-手性拆分) [10]
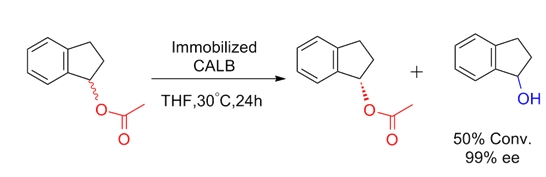
實例 8(酰胺水解反應) [11]
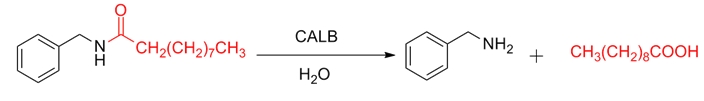
實例 9 (酰胺合成反應) [12]

實例 10(加成反應) [13]

CALB在水相和非水相中都保留著很強的催化活力,無論在水解,酯化,還是酯合成反應等方麵,CALB都表現出了高於(yu) 一般脂肪酶的催化性能,因而在精細化工、藥物拆分以及有機合成方麵有著巨大的潛力。
二、CALB催化機理
CALB的催化機理可以用“乒乓機製”來解釋,主要分為(wei) 兩(liang) 個(ge) 過程,即酰化和去酰化。在酰化過程中,底物1(例如酯類、碳酸鹽等)進入活性位點提供酰基,形成第一個(ge) 四麵體(ti) 中間體(ti) ,釋放出醇類並形成酰基酶。在去酰化過程中,底物2(例如醇類、半縮醛等)進入活性位點作為(wei) 酰基受體(ti) ,生成第二個(ge) 四麵體(ti) 中間體(ti) ,釋放出酯類和自由的CALB。如果反應在水溶液中進行,則底物2為(wei) 水[1,14]。
三、佰利乐生物的CALB固定化酶介紹
佰利乐生物從(cong) 2007年來就專(zhuan) 注於(yu) 生物酶和生物催化技術以及合成生物學技術的開發與(yu) 應用研究。佰利乐生物現有的CALB固定化酶為(wei) CALB通過疏水作用物理吸附於(yu) 聚甲基丙烯酸樹脂上得到的固定化酶,應用於(yu) 水相或有機相反應中,可達到反複套用的效果,套用次數與(yu) 反應體(ti) 係組成有關(guan) 。我司CALB固定化酶產(chan) 品代號SZ-CALB-IMMO100-A和SZ-CALB-IMMO100-B,其中SZ-CALB-IMMO100-A活力可以達到15000U/g。
四、參考文獻
[1] 王亞(ya) 嬋. 南極假絲(si) 酵母脂肪酶B 熱穩定性改造及在黑曲黴中的表達[D]. 無錫:江南大學. 2023.
[2] Uppenberg J, Hansen M T, Patkar S, et al. Structure, 1994, 2(4): 293-308.
[3] J Rotticci-Mulder. Stockholm:Bioteknolog, 2003. 1-74.
[4] Chen S, Liu F, Zhang K, e tal. Tetrahedron Lett, 2016, 57: 5312-5314.
[5] Olah M, Boros Z, anszky G H, e tal. Tetrahedron, 2016, 72: 7249-7255.
[6] Nakaoki1 T, Mei Y, Miller L M, e tal. Ind. Biotechnol, 2005, 1(2):126-134.
[7] Pawar S V, Yadav G D. J. Ind. Eng. Chem, 2015, 31: 335-342.
[8] Kamble M P, Shinde S D, Yadav G D. J. Mol. Catal. B: Enzym, 2016, 132: 61-66.
[9] Shinde S D, Yadav G D. Process Biochem, 2015, 50: 230-236.
[10] Souza T C, Fonseca T S, Costa J A, e tal. J. Mol. Catal. B: Enzym, 2016, 130: 58-69.
[11] Gavil´an A T, Castillo E, L´opez-Mungu´A. J. Mol. Catal. B: Enzym, 2006, 41: 136-140.
[12] Joubioux F L, Henda Y B, Bridiau N, e tal. J. Mol. Catal. B: Enzym, 2013, 85-86: 193-199.
[13] Dhake K P, Tambade P J, Singhal R S, e tal. Tetrahedron Lett, 2010, 51: 4455-4458.
[14] Javier Gonza´lez-Sabı´n, Iva´n Lavandera, e tal. Tetrahedron: Asymmetry, 2006, 17:1264-1274.
搜索


-
-

